2023年02月20日,上海交通大學(xué)醫(yī)學(xué)院上海市免疫學(xué)研究所葉幼瓊研究員團隊在Nature Communications 上發(fā)表題為“Reconstruction of the tumor spatial microenvironment along the malignant-boundary-nonmalignant axis”的研究論文。該研究開發(fā)了腫瘤空間微環(huán)境解析工具Cottrazm (Construction of Tumor Transition Zone Microenvironment), 結(jié)合空間轉(zhuǎn)錄組數(shù)據(jù)和HE染色圖像以及單細胞轉(zhuǎn)錄組數(shù)據(jù),精準(zhǔn)繪制了腫瘤組織中連接惡性和非惡性區(qū)域的腫瘤邊界區(qū)域,利用反卷積的方法解析不同空間定位上的細胞組成,并在sub-spot水平重構(gòu)細胞類型特異的基因表達譜。利用該工具以結(jié)直腸癌和肝癌的腫瘤樣本為例,發(fā)現(xiàn)在腫瘤邊界富集的特定細胞亞型、細胞互作、以及靶向邊界的潛在治療靶點,為臨床靶向腫瘤空間微環(huán)境治療提供了新策略。
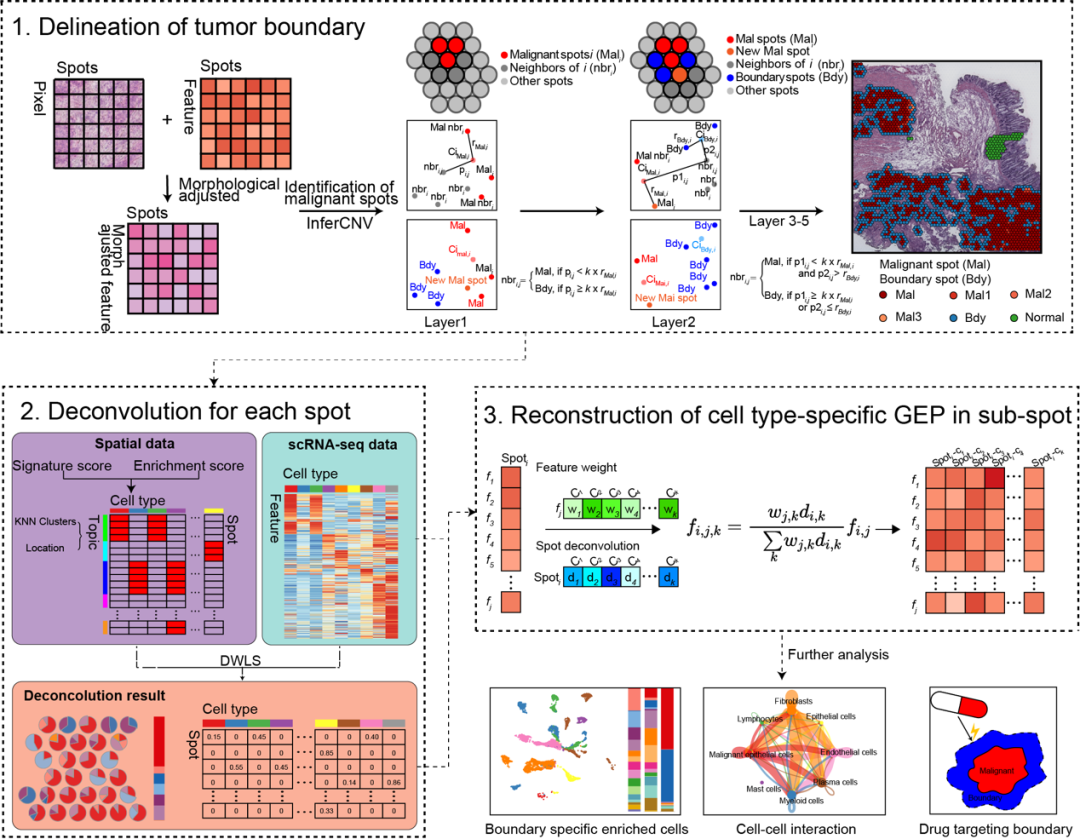
腫瘤邊界是由位于實體瘤最外層的惡性細胞和在空間結(jié)構(gòu)上緊密相連的非惡性細胞構(gòu)成的、連接惡性和-非惡性空間組織的橋梁。盡管腫瘤邊界的細胞組成、細胞相互作用和分子網(wǎng)絡(luò)調(diào)控都對腫瘤微環(huán)境(Tumor microenvironment, TME)重塑有重要影響,但腫瘤邊界的概念通常是由科學(xué)家或病理學(xué)家基于IHC染色數(shù)據(jù)進行主觀描述,長期以來缺乏明確的標(biāo)準(zhǔn)或一致的方法[1]。
空間轉(zhuǎn)錄組 (Spatial transcriptomics, ST) 結(jié)合單細胞轉(zhuǎn)錄組測序 (single-cell RNA sequencing, scRNA-seq) 的優(yōu)勢在于可以直接在組織上賦予基于表達譜二維空間信息。大多原位捕獲技術(shù)諸如10X Genomics Visium, 往往在一個空間點中包含多個同質(zhì)或異質(zhì)細胞,使得準(zhǔn)確定義空間點的細胞組成具有挑戰(zhàn)性。傳統(tǒng)的ST分析工具通常聚焦于圖像分析、細胞類型定義、反卷積、空間分布、細胞間相互作用和空間基因表達模式等[2-5],目前尚無針對腫瘤邊界的分析工具,很大程度上限制了對腫瘤邊界的細胞組成、邊界中不同類型細胞間相互作用以及基于腫瘤邊界的潛在治療靶點的研究。
葉幼瓊研究團隊開發(fā)了腫瘤空間微環(huán)境解析工具Cottrazm,一個基于10x Genomics Visium平臺ST測序構(gòu)建腫瘤邊界周圍微環(huán)境的R包 (圖1)。本項研究中首先利用Cottrazm界定了連接惡性和非惡性空間點的腫瘤邊界 (Cottrazm-BoundaryDefine);其次通過整合scRNA-seq、空間轉(zhuǎn)錄組學(xué)和空間點的位置信息通過反卷積算法預(yù)測空間各點的細胞組成 (Cottrazm-SpatialDecon);最終利用ST和scRNA-seq數(shù)據(jù)在sub-spot水平重建了細胞類型特異的基因表達譜 (Cottrazm-SpatialRecon)。
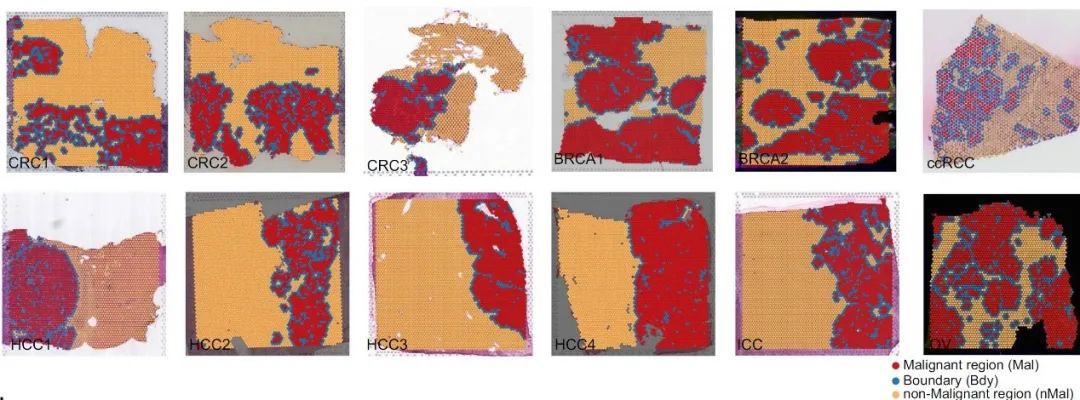
研究進一步利用ST模擬數(shù)據(jù)驗證了Cottrazm在空間點的細胞浸潤預(yù)測和空間sub-spot的細胞類型特異性基因表達譜的預(yù)測上具有較高的準(zhǔn)確性和敏感性。研究團隊利用Cottrazm分析了6種冰凍腫瘤組織的13個ST數(shù)據(jù)集和2種福爾馬林固定石蠟包埋 (FFPE) 癌癥組織的3個ST數(shù)據(jù)集, 結(jié)果顯示所有樣本均可勾勒出清晰的腫瘤邊界從而區(qū)分惡性區(qū)域和非惡性區(qū)域(圖2)。同時以結(jié)直腸癌(Colorectal Cancer,CRC)為例,反卷積預(yù)測CRC樣本中每個空間點(spot)的細胞組成,三個區(qū)域的細胞組成顯示絕大多數(shù)惡性spot主要由腫瘤細胞組成,缺少T細胞或其他類型細胞的浸潤;邊界spot主要由髓系細胞,成纖維細胞和內(nèi)皮細胞組成;非惡性spot中不含腫瘤細胞且在不同組織區(qū)域細胞組分特征不同。為進一步解析CRC空間微環(huán)境,本研究重構(gòu)了每個spot中各亞類細胞(sub-spot)的基因表達圖譜(Gene Expression Profiles, GEPs),并在sub-spot水平進行細胞亞群重注釋(圖3)。深入分析顯示,具有腫瘤免疫排斥型微環(huán)境的CRC和肝癌(HepatoCellular Carcinoma,HCC)中髓系細胞亞群Macro-SPP1和成纖維細胞亞群Fib-ASPN在腫瘤邊界富集并和惡性上皮細胞相互作用形成屏障,阻礙T細胞在惡性區(qū)域的浸潤,為未來靶向腫瘤邊界的治療篩選了潛在靶點。
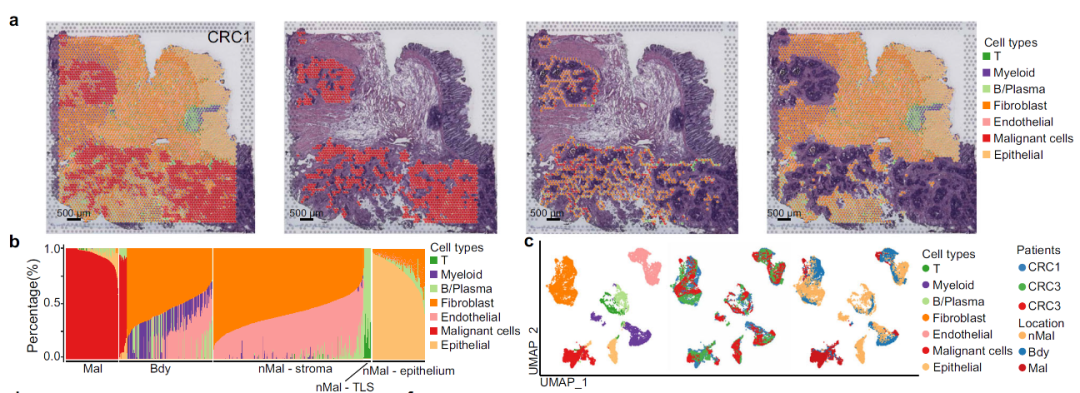
圖2. Cottrazm精準(zhǔn)繪制不同癌種中惡性腫瘤區(qū)域(紅色)、腫瘤邊界區(qū)域(藍色)和非惡性區(qū)域(橙色)

圖3. Cottrazm預(yù)測腫瘤空間微環(huán)境中不同區(qū)域的細胞組成和對應(yīng)細胞的表達圖譜
綜上,新開發(fā)的 Cottrazm工具為ST數(shù)據(jù)依賴的TSME解析提供了基于惡性-邊界-非惡性軸的新方法,可用于腫瘤邊界的空間位置、細胞組成、基因表達譜、生物學(xué)功能、細胞相互作用分析和潛在治療靶點的挖掘。從臨床轉(zhuǎn)化的角度來看,利用該工具以結(jié)直腸癌和肝癌的腫瘤樣本為例,發(fā)現(xiàn)在腫瘤邊界富集的特定細胞亞型、細胞互作、以及靶向邊界的潛在治療靶點,該工具可以推廣到泛癌中使用,為臨床靶向腫瘤空間微環(huán)境治療提供了新治療策略。
上海市免疫學(xué)研究所葉幼瓊研究員和美國Texas A&M大學(xué)韓冷教授為該文的共同通訊作者, 上海市免疫學(xué)研究所博士研究生荀臻臻為本論文的獨立第一作者。該研究得到了上海市免疫學(xué)研究所蘇冰教授在空間邊界微環(huán)境概念構(gòu)思等方面的大力支持。該工作受國家自然科學(xué)基金、上海市科學(xué)技術(shù)委員會先導(dǎo)項目的支持。同時感謝上海市免疫學(xué)研究所測序平臺的支持。
 葉幼瓊,研究員,博士生導(dǎo)師,上海交通大學(xué)醫(yī)學(xué)院/上海市免疫學(xué)研究所課題組長(PI)(http://www.yelab.site/)。課題組圍繞腫瘤(邊界)微環(huán)境,以生物大數(shù)據(jù)為驅(qū)動,構(gòu)建算法、開發(fā)工具,結(jié)合生物信息分析、基礎(chǔ)實驗、臨床隊列研究等闡釋腫瘤邊界微環(huán)境中重要調(diào)控因子對腫瘤進展及免疫逃逸的作用機制;并利用多維數(shù)據(jù)構(gòu)建免疫治療療效預(yù)測模型,為免疫治療研究提供新思路新策略。相關(guān)研究成果以最后(共同)通訊作者、第一(共同)作者發(fā)表在Nature Metabolism (2019)、Nature Immunology (2019)、Cancer Cell (2020)、Nature Communications(2020,2022,2023)、Cancer Research (2023)等期刊。葉幼瓊課題組長期招收博士后,具有生物信息學(xué)和腫瘤免疫學(xué)等相關(guān)背景優(yōu)先,歡迎更多優(yōu)秀的人才(包括博士后、助理研究員、技術(shù)員)加入。請感興趣的申請者來函咨詢:[email protected]。
葉幼瓊,研究員,博士生導(dǎo)師,上海交通大學(xué)醫(yī)學(xué)院/上海市免疫學(xué)研究所課題組長(PI)(http://www.yelab.site/)。課題組圍繞腫瘤(邊界)微環(huán)境,以生物大數(shù)據(jù)為驅(qū)動,構(gòu)建算法、開發(fā)工具,結(jié)合生物信息分析、基礎(chǔ)實驗、臨床隊列研究等闡釋腫瘤邊界微環(huán)境中重要調(diào)控因子對腫瘤進展及免疫逃逸的作用機制;并利用多維數(shù)據(jù)構(gòu)建免疫治療療效預(yù)測模型,為免疫治療研究提供新思路新策略。相關(guān)研究成果以最后(共同)通訊作者、第一(共同)作者發(fā)表在Nature Metabolism (2019)、Nature Immunology (2019)、Cancer Cell (2020)、Nature Communications(2020,2022,2023)、Cancer Research (2023)等期刊。葉幼瓊課題組長期招收博士后,具有生物信息學(xué)和腫瘤免疫學(xué)等相關(guān)背景優(yōu)先,歡迎更多優(yōu)秀的人才(包括博士后、助理研究員、技術(shù)員)加入。請感興趣的申請者來函咨詢:[email protected]。
【參考文獻】
1. Ji, A. L. et al. Multimodal Analysis of Composition and Spatial Architecture in Human Squamous Cell Carcinoma. Cell 497–514 (2020).
2. Rao, A., Barkley, D., França, G. S. & Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature 596, 211–220 (2021).
3. Asp, M., Bergenstråhle, J. & Lundeberg, J. Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration. BioEssays 42, 1–16 (2020).
4. Liao, J., Lu, X., Shao, X., Zhu, L. & Fan, X. Uncovering an Organ’s Molecular Architecture at Single-Cell Resolution by Spatially Resolved Transcriptomics. Trends Biotechnol. 39, 43–58 (2021).
5. Ståhl, P. L. et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science vol. 353 78–82 (2016).
 滬公網(wǎng)安備 31009102000053號 滬ICP備18007527號-1 郵箱:[email protected]
滬公網(wǎng)安備 31009102000053號 滬ICP備18007527號-1 郵箱:[email protected] 滬公網(wǎng)安備 31009102000053號 滬ICP備18007527號-1 郵箱:[email protected]
滬公網(wǎng)安備 31009102000053號 滬ICP備18007527號-1 郵箱:[email protected]